迄今最大合成基因组诞生:非全部密码子构建的
迄今为止最大的合成基因组诞生了,这个只有部分氨基酸编码密码子的基因组让未来编码出含有非天然氨基酸残基的蛋白成为了可能。
过去十年来,随着DNA化学合成的成本不断降低,DNA片段的组装方式日益精进,合成生物学迈入了合成整个染色体和基因组的新阶段。迄今为止,研究人员可以用最多100万碱基对构建出合成DNA,比如之前酿酒酵母(Saccharomyces cerevisiae )的一组染色体以及丝状支原体(Mycoplasma mycoides)的各种合成基因组。如今,Fredens等人在《自然》上发表论文称,团队合成了一个有400万碱基对的大肠杆菌(Escherichia coli)基因组。该研究是合成基因组学这一新兴领域内具有里程碑意义的事件,也是合成基因组技术在这个“劳苦功高”的细菌上的首次应用。

合成基因组学赋予了我们认识生命规则的全新方式,同时也在推动合成生物学朝着基因组编写设计的方向前进。这一领域的先驱是来自美国克雷格·文特 尔研究所(J。 Craig Venter Institute)的研究人员,为了确定独立生存细胞所需的最少基因数量,他们选择通过这种方法先让计算机对基因组片段进行重新设计,再化学合成这些片 段,最后完成组装。凭借这一技术,这些研究人员成功地将丝状支原体的基因组大小缩减了约50%。如果换用基因组编辑工具则会大大增加工作量,以往的大肠杆 菌实验已经证实了这一点——基因删除方法最多只能去除15%的基因组。
Fredens和同事将缩小后的大肠杆菌基因组作为一个合成基因组模板,但他们脑中还有另一种最小化的办法——缩减密码子。遗传密码本身具有冗 余性:一共有64个密码子(碱基三联体)编码20种氨基酸,以及代表一段蛋白编码序列启动和终止的“起始”点和“终止”点。这种冗余意味着有6个编码丝氨 酸的密码子和3个可能的终止密码子。大肠杆菌的蛋白编码序列有64个可用密码子,Fredens等人通过设计、合成和组装,只用61个密码子就构建出了大 肠杆菌基因组,他们的做法是将两个丝氨酸密码子和一个终止密码子用同义密码子替代——同义密码子是指“拼写”不同但发出指令相同的密码子。之前使用基因组 编辑工具的研究成功用63个密码子合成了一个大肠杆菌,但这只需要将序列为TAG的终止密码子(基因组中有321个)替换成另一个终止密码子。而这次缩减 至61个密码子则需要改变18214个密码子,为此必须采用基因组合成的方法。
Fredens和同事通过之前为研究大肠杆菌密码子缩减极限而开发的大规模DNA组装和基因组整合方法,合成了这个大肠杆菌基因组。他们先在计 算机上对DNA进行设计,再在酿酒酵母载体中化学合成并组装100千碱基片段,这些载体随即被大肠杆菌摄取,并直接整合到基因组中对应的天然区域(见图 1)。将这个过程迭代5次,就能让500千碱基的DNA节段完全被合成DNA取代。研究团队用这种方法生成了8个大肠杆菌菌株,每个菌株都携带有覆盖不同 基因组区域的合成DNA节段。随后,研究人员再通过“接合”的方法将这些节段拼凑起来,合成完整的基因组。
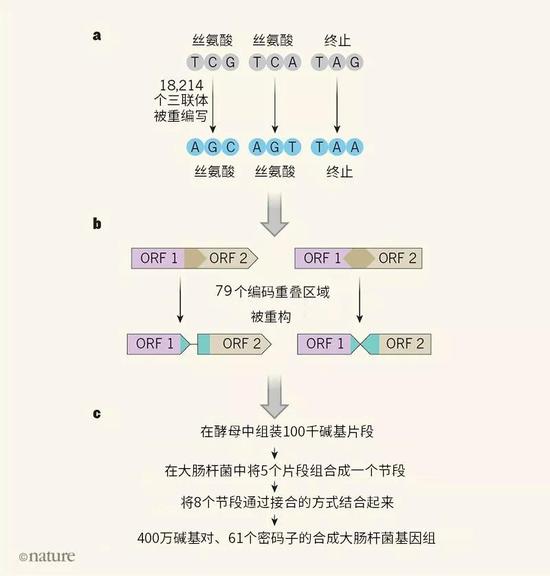
b、在基因组的某些位点上,开放读码框(ORF,即蛋白编码区域)会发生重叠,对某个ORF密码子做出改变可能会导致重叠区域出现非预期的变 化。Fredens等人通过“重构”这些ORF使其分离,如图中的ORF1和ORF2(左边两个ORF的“读取”方向相同,右边两个读取方向相反)。
c、经过重新设计的DNA可以在酿酒酵母中合成组装100千碱基片段;这些片段再组合成节段,整合到大肠杆菌的基因组中。将这些节段结合就能获得完整的功能合成基因组。
这一大规模的构建工程取得了惊人的成功,脱靶突变率很低,但也存在一些挑战。大肠杆菌基因组中的许多基因会与其它基因有部分重叠,研究人员发现 有91个存在重叠区域含有需要改变的密码子的情况。这很复杂,因为一个蛋白编码序列中的同义替换可能会使重叠序列编码的氨基酸发生改变。为了解决这个问 题,研究团队对基因组中的79个位点进行了“重构”,通过复制这段序列,他们将重叠的编码序列分开,变成单个重编码序列(见图1)。虽然这一方法基本上很 成功, 但有些地方需要非常小心的错误排查,因为重构可能改变了基因调控。
最后得到的菌株被证实可以存活,并能在各种典型的实验室环境下生长,但是比天然菌株长得稍慢一些。新的菌株不再需要终止密码子TAG以及丝氨酸 密码子TCG和TCA,因此,识别这些密码子的细胞机器也可以被去除或重新用于招募“非标准”氨基酸——大部分活细胞所需的20种常见氨基酸之外的氨基 酸。研究人员用只有63个密码子的大肠杆菌证实了招募非标准氨基酸的用处。对于生物技术项目来说,非标准氨基酸可以被写入理想的序列位置,提供可以参与天 然蛋白无法参与的化学反应的残基。不仅如此,由于支持合成大肠杆菌细胞运作的遗传密码稍不同于自然世界中的其它细胞,输入和输出合成大肠杆菌的可读取 DNA编码信息将变得有限,并因此带来了生物安全方面的益处。以上种种应用都会在新的61个密码子的大肠杆菌中得到进一步拓展,新菌株有潜力编码不止一个 非标准氨基酸的使用,并建立一个更严苛的基因防火墙(因为64个密码子中的3个密码子已不再被识别)。
合成400万碱基对的基因组,以及将遗传密码缩减至61个密码子创造了合成基因组学的最新纪录,但这个纪录可能不会保持得太久。国际Sc2.0 联盟正在设法合成有1200万碱基对的酿酒酵母基因组的全部16个染色体,这也是首个真核生物(包括植物、动物和真菌)的合成基因组。同时,联盟还在尝试 合成只有57个密码子的大肠杆菌基因组以及比天然鼠伤寒沙门氏菌(Salmonella Typhimurium)少两个密码子的合成基因组。如果成功,将来拥有合成基因组的细菌有望作为基于细胞的技术,应用于人体肠道。
从纯技术的角度来说,这些不同的研究最值得关注的点在于它们构建合成基因组的流程极为相似,都是将合成DNA的千碱基节段(通过同源重组)在酵 母细胞中组装成 50-100千碱基片段,再用这些片段(通过选择重组的方法)替代目标生物体中的天然序列。方法的标准化有利于实现部分步骤的自动化,让更多团队加入研究 行列。基因组最小化和密码子缩减只是这一技术的初步应用,今后,这种技术或能生成功能重组基因组,或者能指示细胞执行特定任务的“定制”基因组。